PythonによるCSVファイル取り込み
PythonによるCSVデータの取り込み
NIDD 国立感染症研究所
全数把握疾患、報告数、累積報告数、都道府県別
サンプルにするデータはIDWR速報データの五類感染症に関して都道府県別発生件数を週ごとにまとめられたCSVファイルを使用します。
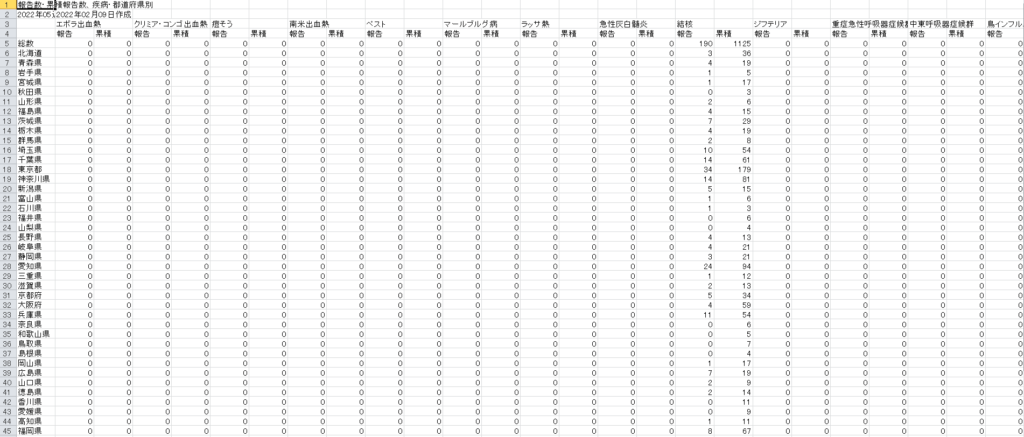
発生頻度が0のところは見にくいのでこういった部分を省いてデータを集約して見やすくし、さらには週ごとのデータも集約する方法になります。
Jupyter Lab
今回はAnaconda3のJupyter Lab上で操作を進めていくことにします。同じanaconda3上にjupyternotebookというのがありますが現在は開発が止まっているのでjupyter labのほうですすめていくほうが無難でしょう。

集計CSVデータのダウンロード
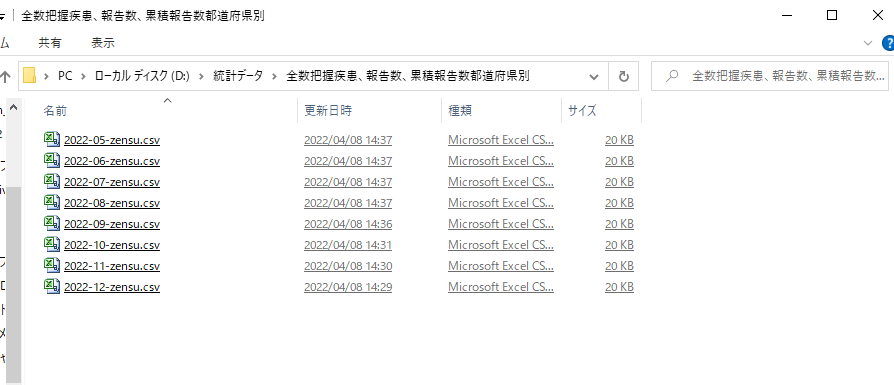
以下のリンクからデータをダウンロード。
その他2022年第6週から第13週は右サイドバーの一番下のリンクが貼ってあるのでそれぞれをダウンロードして適当なフォルダに入れておきます。
pandasのインポート
jupyter labを開いたら適当なファイル名をつけてpandasとglobのインポートを実施。
import pandas as pd from glob import glob
次にglobでファイルのあるパスを指定して読みこませます(.csvの前にある*はワイルドカード)。
glob ('D:\統計データ\全数把握疾患、報告数、累積報告数都道府県別/*.csv')
※)パスの指定がわからない場合はファイルのある所に直接入っていって次のようにパスをそのまま最初の’と*の間にコピペします。
Shift + Enterで実行させて次のようにファイルを読み込ませる。

それを新しくzensu_haakuの中に入れる。
zensu_haaku = glob ('D:\統計データ\全数把握疾患、報告数、累積報告数都道府県別/*.csv')
zensu_haaku

0番目のファイルをdir_pathへ格納します。
dir_path = zensu_haaku[0] dir_path
仮置きの中にpd.read_csvでファイル読み込みさせる。
df_temp = pd.read_csv(dir_path, encoding='sjis') df_temp
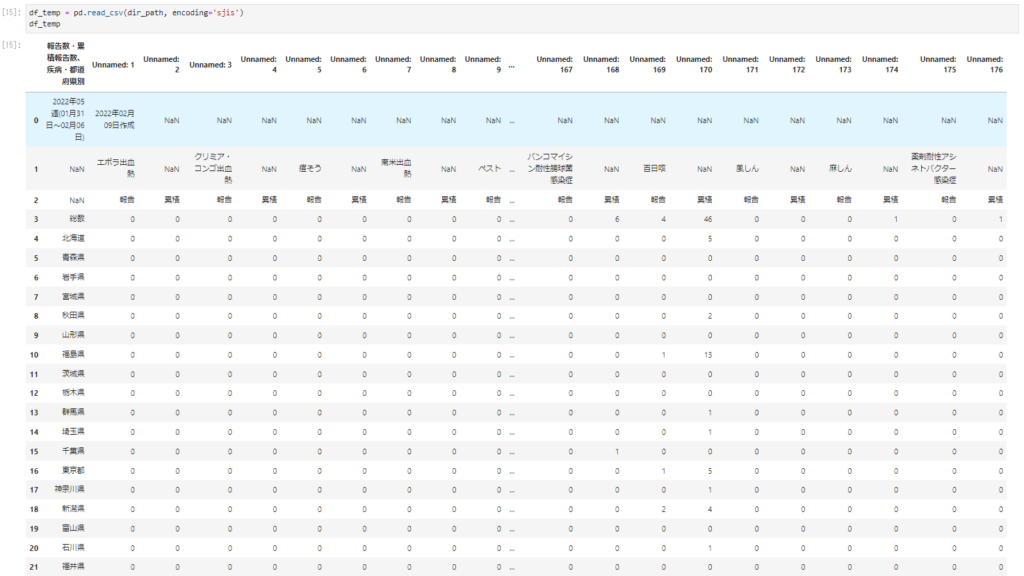
主にデータの入っている箇所の列と、なんの症例か判別するために主要感染症名のある行を取り出してcolumnsに入れておきます。
columns = df_temp.iloc[1, [0, 17, 35, 41,45,55,81,93,123,129,131,133,137,141,143,145,149,153,159,161,163,169]] columns
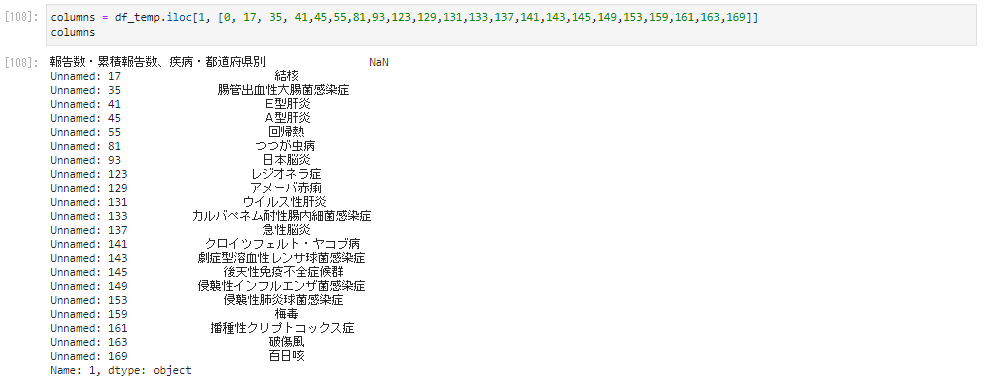
右上に欠損値のNaNがあるのでここを“都道府県別”という文字列を代入します。
次のように入力。
columns = columns.fillna('都道府県別')
columns

次にデータの入っている都道府県別の行を取り出していく。
df = df_temp.iloc[4:50, [0, 17, 35, 41,45,55,81,93,123,129,131,133,137,141,143,145,149,153,159,161,163,169]] df
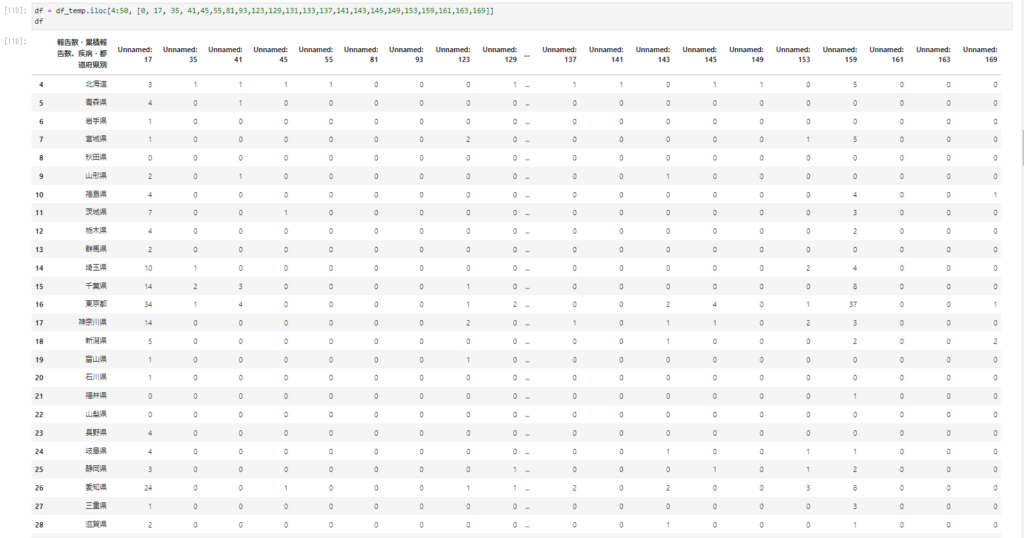
なんの列名(病名)かわかるように先ほど作成したcolumnsを入れてやるようにする。
df.columns = columns df

いつの時期のものかわかるように週数を入れる。
df['週数'] = df_temp.iloc[0, 0] df
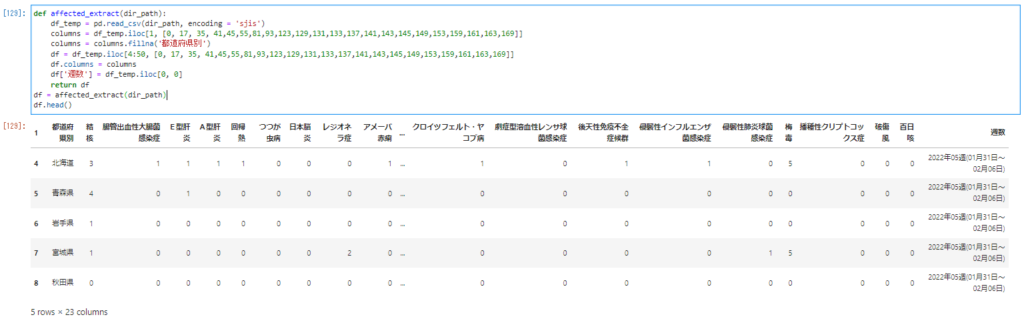
一連のデータ抽出の流れをまとめてaffected_extractという関数にまとめます。ファイルのパスを認識させるために関数名の後の()の中にdir_pathと入力してファイルを引き込んでやります。
def affected_extract(dir_path):
df_temp = pd.read_csv(dir_path, encoding = 'sjis')
columns = df_temp.iloc[1, [0, 17, 35, 41,45,55,81,93,123,129,131,133,137,141,143,145,149,153,159,161,163,169]]
columns = columns.fillna('都道府県別')
df = df_temp.iloc[4:50, [0, 17, 35, 41,45,55,81,93,123,129,131,133,137,141,143,145,149,153,159,161,163,169]]
df.columns = columns
df['週数'] = df_temp.iloc[0, 0]
return df
df = affected_extract(dir_path)
df.head()

データの集約
pd.concat
抽出のコードが出来たので週ごとのデータをひとつのデータにまとめます。
df = pd.DataFrame() for dir_path in zensu_haaku: df_temp = affected_extract(dir_path) df = pd.concat([df, df_temp]) df
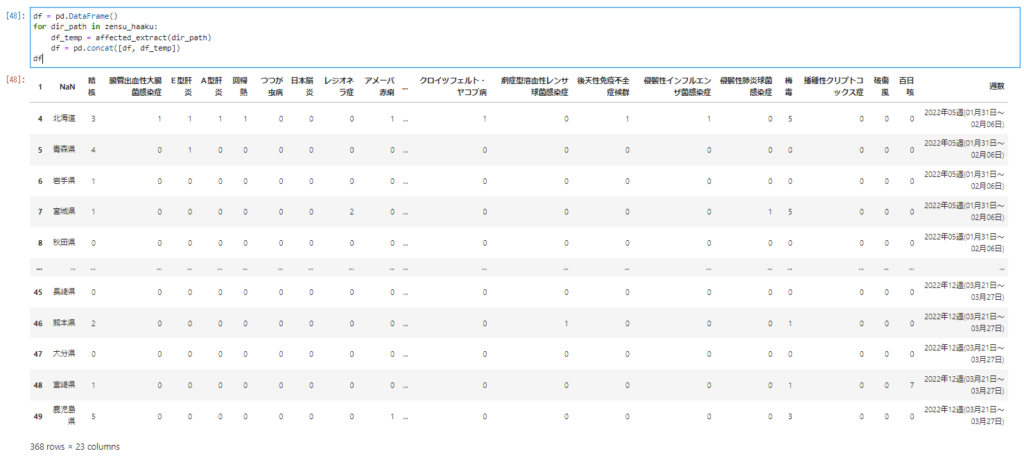
左下の行数を見ればわかるように368 rows × 23 columnsとなっているのでデータが集約出来ていることがわかります。
左の行番を順通りにするために以下のように入力。
df = df.reset_index(drop = True)
次にこれをCSVファイルとして出力します。
df.to_csv('zensu_data.csv', index = False)
実行するとzensu_haku.csvという名前のファイルが作られます。
これをダブルクリックしてやればjupyter lab上で以下のように作成されたファイルが閲覧出来るようになります。
CSVファイルの文字化け修正
なおカレントディレクトリから開く場合csvファイルが以下のように文字化けしていることがあります。

次のように対処していきます。
まず対象の文字化けしているCSVを右クリックしてそれをメモ帳か何かで開きます。
名前を付けて保存をクリック。

この時文字コードをANSIに変換して上書き保存。
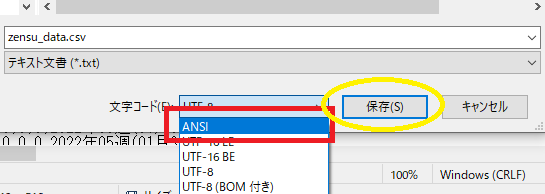
文字コードをANSIにして上書き保存します。

再度開き直せば次のように表示されるようになります。
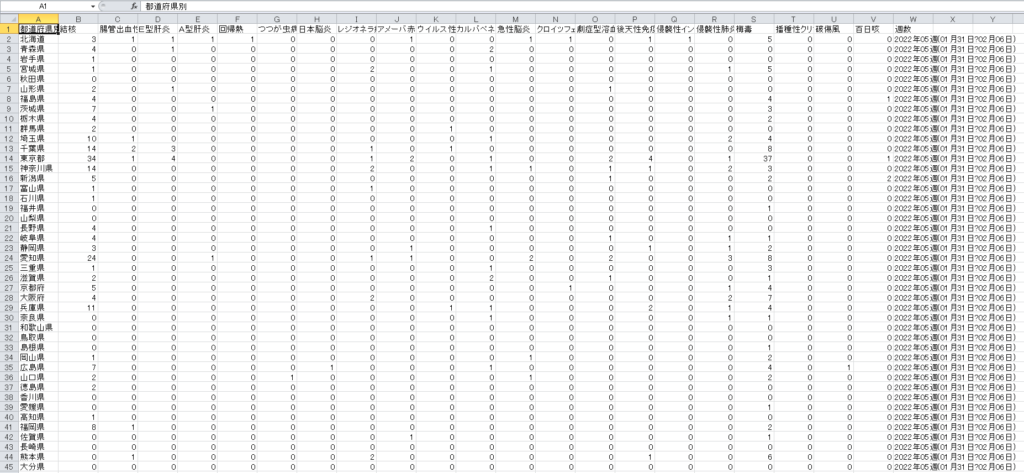
出力
都道府県ごとのデータを出力してexport_dirというフォルダに格納します。
for i in prefectures:
df_affected = df[df['都道府県別'] == i]
df_affected.to_csv('D:\統計データ\全数把握疾患、報告数、累積報告数都道府県別\export_dir' + '/' + i + '.csv')
glob ('D:\統計データ\全数把握疾患、報告数、累積報告数都道府県別\export_dir/*.csv')
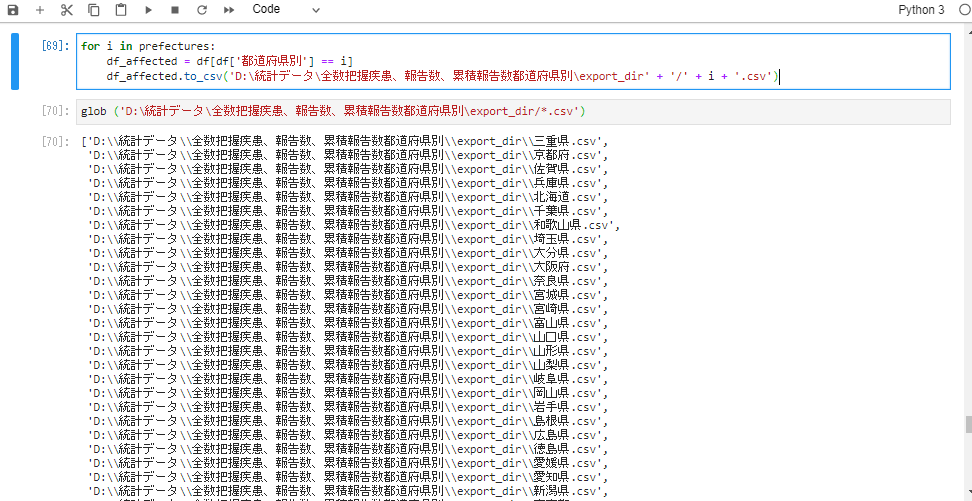
all_csv_dataへ格納します。
all_csv_data = glob ('D:\統計データ\全数把握疾患、報告数、累積報告数都道府県別\export_dir/*.csv')
all_csv_data
for文のなかにconcatを使ってdf_all_csv_dataへデータを結合します。
df_all_csv_data = pd.DataFrame() for i in all_csv_data: df_read_csv = pd.read_csv(i) df_all_csv_data = pd.concat([df_read_csv, df_all_csv_data])
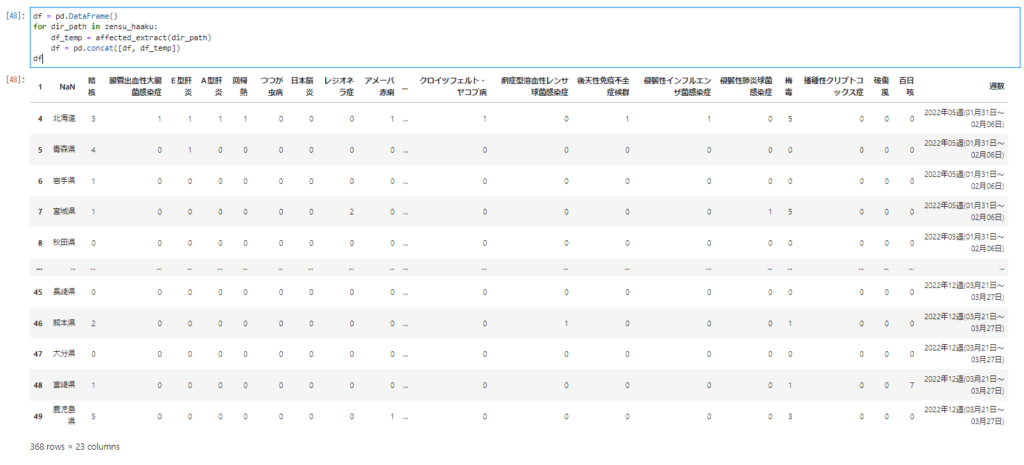
余計な列があるのでこれを次のようにして削除します。
df_drop = df_all_csv_data.drop('Unnamed: 0', axis = 1)
df_all_csv_data = df_drop
df_all_csv_data
次のように入力して行番をリセット。
df_all_csv_data = df_all_csv_data.reset_index(drop = True) df_all_csv_data
最後に次のようにしてCSVデータを出力。
df_all_csv_data.to_csv('all_csv_data.csv', index = False)

データの描画
seabornの日本語対応をしていないのでそれぞれに対応する病名を次のように表記。
| 結核 | tuberculosis |
| 腸管出血性大腸菌感染症 | EHEC(Enterohemorrhagic Escherichia coli infection) |
| E型肝炎 | Hepatitis E |
| A型肝炎 | Hepatitis A |
| 回帰熱 | recurrent fever |
| つつが虫病 | Tsutsugamushi disease |
| 日本脳炎 | Japanese encephalitis |
| レジオネラ症 | Legionellosis |
| アメーバ赤痢 | Amoebiasis |
| ウイルス性肝炎 | Viral hepatitis |
| カルバペネム耐性腸内細菌感染症 | CRE(carbapenem-resistant Enterobacteriaceae) |
| 急性脳炎 | Acute encephalitis |
| クロイツフェルト・ヤコブ病 | CJD(Creutzfeldt-Jakob disease) |
| 劇症型溶血性レンサ球菌感染症 | Severe invasive streptococcal disease |
| 後天性免疫不全症候群 | HIV |
| 侵襲性インフルエンザ菌感染症 | Invasive Haemophilus influenzae disease |
| 侵襲性肺炎球菌感染症 | invasive pneumococcal disease |
| 梅毒 | Syphilis |
| 播種性クリプトコックス症 | Cryptococcosis |
| 破傷風 | tetanus |
| 百日咳 | Whooping cough |
df_prefectures_master.index = ['mie', 'kyoto', 'saga', 'hyogo', 'hokkaido', 'tiba', 'wakayama', 'saitama', 'ooita', 'oosaka', 'nara', 'miyagi', 'miyazaki', 'toyama', 'yamaguti', 'yamagata', 'yamanasi', 'gifu', 'okayama', 'iwate', 'tottori', 'hirosima', 'tokusima', 'ehime', 'aiti', 'niigata', 'tokyo', 'totigi', 'siga','kumamoto', 'isikawa', 'kanagawa', 'fukui', 'fukuoka', 'fukusima', 'akita', 'gunma', 'ibaraki', 'nagasaki', 'nagano', 'aomori', 'sizuoka', 'kagawa', 'kouti', 'tottori', 'kagosima' ] df_prefectures_master.columns = ['tuberculosis', 'EHEC', 'Hepatitis E', 'Hepatitis A', 'recurrent fever', 'Tsutsugamushi disease', 'Japanese encephalitis', 'Legionellosis', 'Amoebiasis', 'Viral hepatitis', 'CRE', 'Acute encephalitis', 'CJD', 'Severe invasive streptococcal disease', 'HIV', 'Invasive Haemophilus influenzae disease', 'invasive pneumococcal disease', 'Syphilis', 'Cryptococcosis', 'tetanus', 'Whooping cough'] df_prefectures_master

seabornのインポート
次のように入力。
import matplotlib.pyplot as plt %matplotlib inline import seaborn as sns
都道府県ごとの疾患別感染状況をわかりやすくするためにヒートマップでCSVの数値データを可視化。
次のように入力。
plt.figure(figsize = (15, 6))
cmap = sns.light_palette("#00008b", as_cmap = True)
sns.heatmap(df_prefectures_master, cmap = cmap)
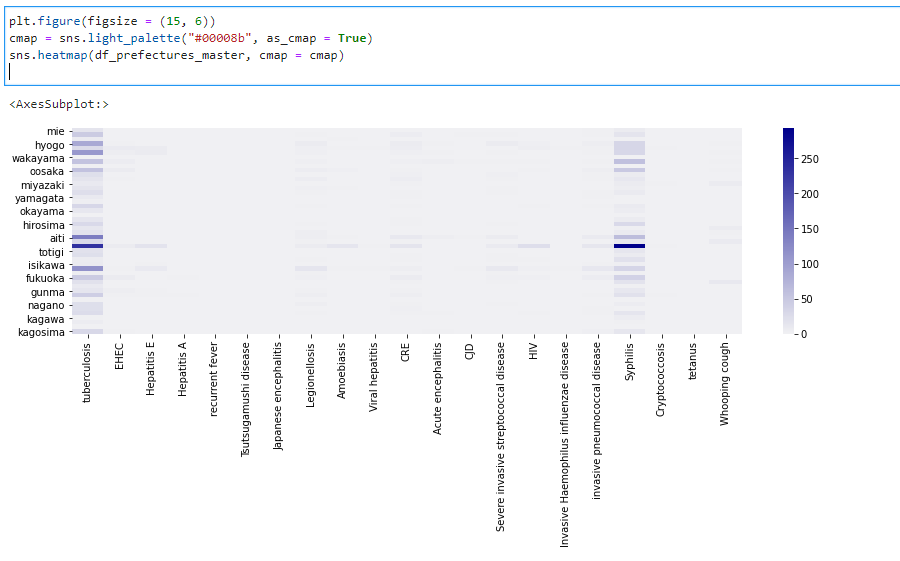
一番左側の疾患が結核で右から4番目の濃い色の列が梅毒になる。
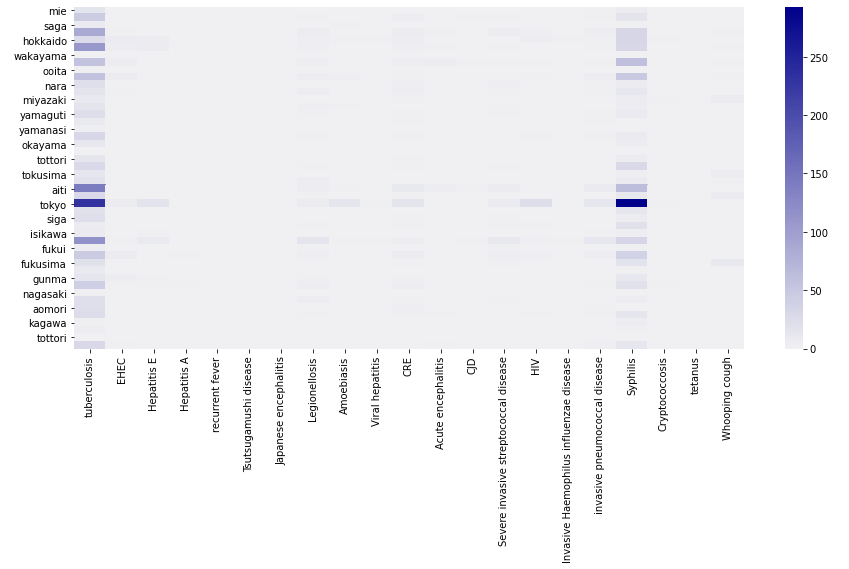
一部の地域を除いて梅毒が密集人口の高いところで頻発しておりとりわけと東京での結核と梅毒感染者が多いことがわかります。
-
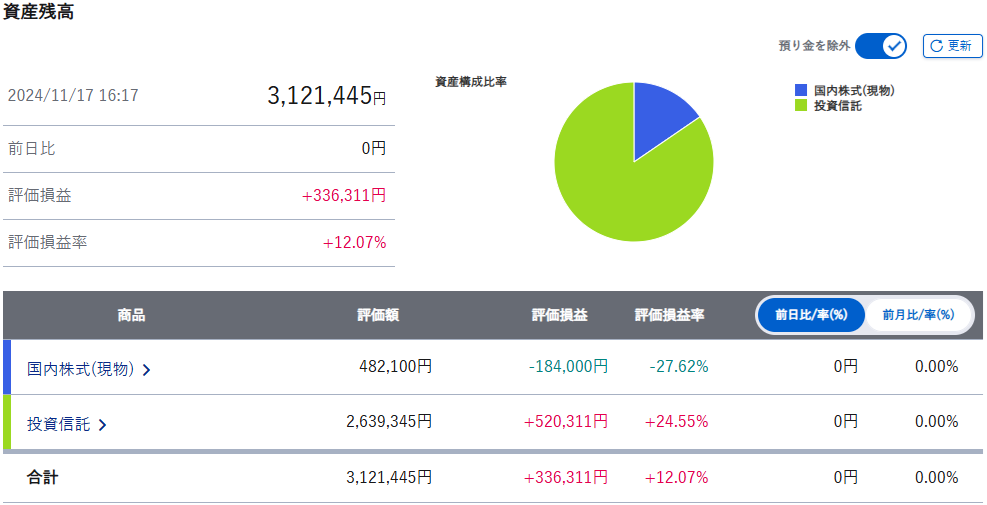
2024年NISA運用概況
カテゴリー
-
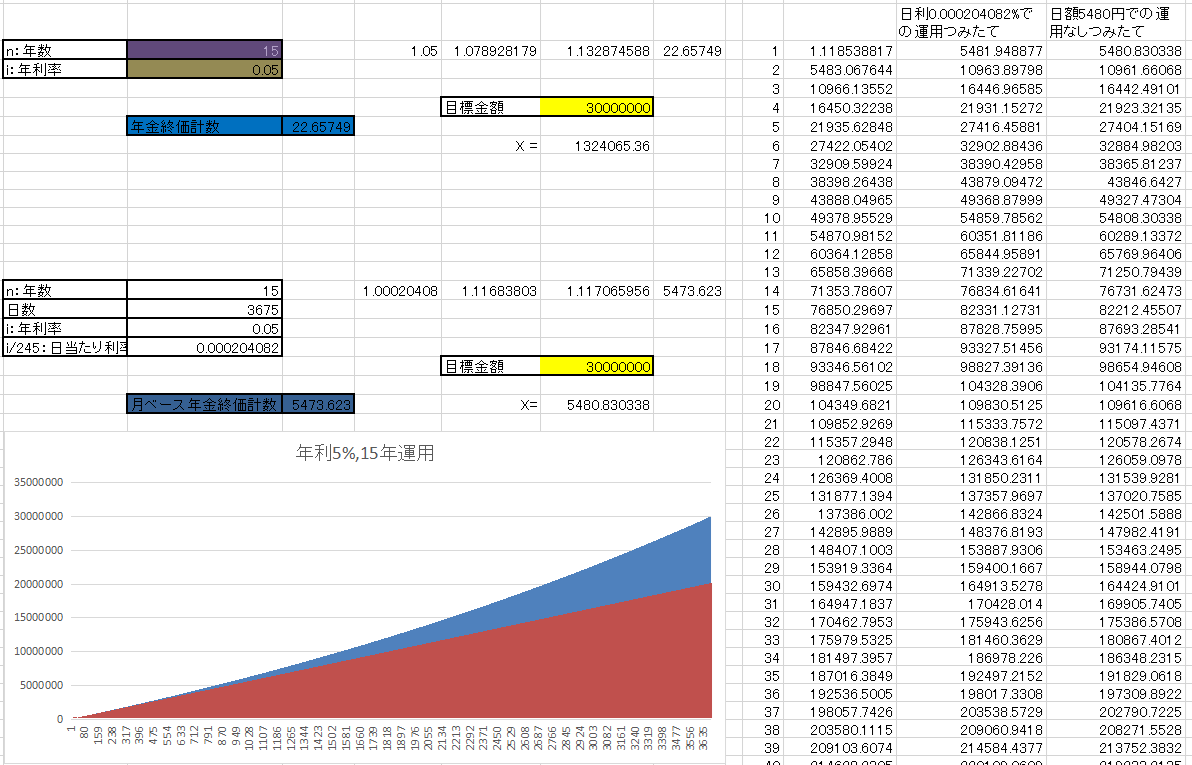
NISA運用概況
カテゴリー
-
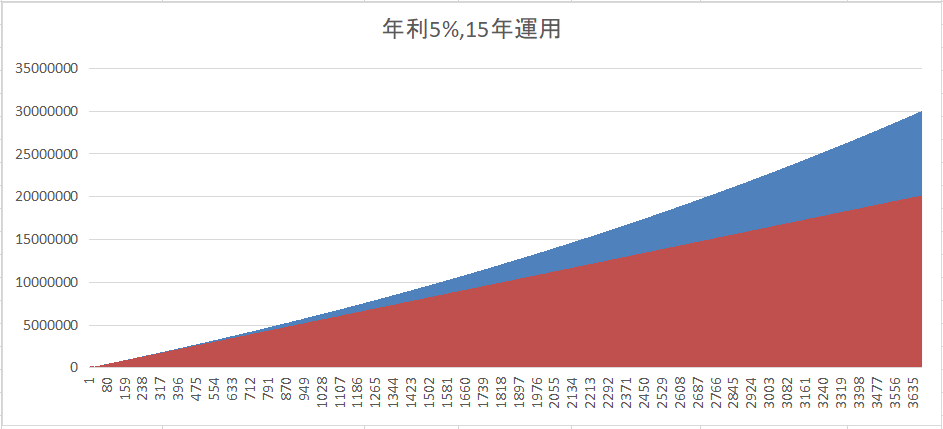
アッパーマス層と年金終価係数
カテゴリー
-
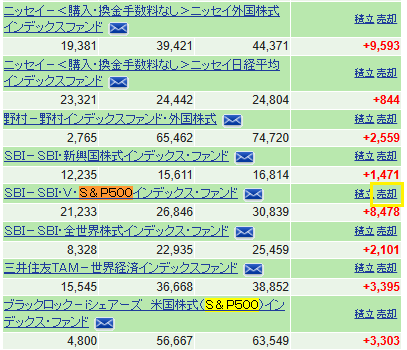
重複分散適宜売却型日別ドルコスト平均法
カテゴリー
-
ネットワーキングラボ
カテゴリー
-
RstudioでSIRモデル②
カテゴリー
テスト投稿
-

2024年NISA運用概況
続きを読む
-

NISA運用概況
続きを読む
-

アッパーマス層と年金終価係数
続きを読む
-

重複分散適宜売却型日別ドルコスト平均法
続きを読む
-

ネットワーキングラボ
続きを読む
-

RstudioでSIRモデル②
続きを読む
-
PythonによるCSV取り込み
カテゴリー : PythonによるCSVファイル取り込みサンプルにするデータはIDWR速報データの五類感染症に関して都道府県別発生件数を週ごとにまとめられたCSVファ…